Lieber Udo,
herzlichen Dank für die ausführliche Lösung zu meiner Frage.
Erlaube bitte eine ergänzende Frage, da es mir leider nicht gelang, den
Code so abzuändern dass statt
[image: image.png]
nur
[image: image.png]
das als Kante im Graph erscheint.
Freundliche Grüße sendet
Peter
Am Di., 25. Aug. 2020 um 22:09 Uhr schrieb Susanne & Udo Krause <
su.krause@XXXXXXX.ch>:
> Guten Abend Peter,
>
> die Wand verschwindet wie Nebel am späteren Vormitttag, denn die
> EdgeLabels müssten stets an eine Kante {isotope1, isotope2} angetragen
> werden --- und nicht an einen Punkt (im RelationGraph ist jedes erreichbare
> Isotop eine Punkt) --- und müssen bei RelationGraph[] im Voraus berechnet
> werden; also unter Verwendung Ihrer Funktionen
>
>
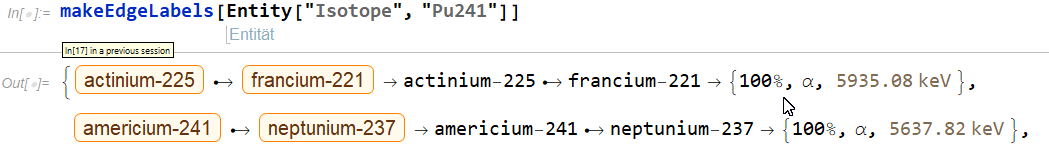
> Clear[decaytypelist, GetEdgeLabel, makeEdgeLabels]
> decaytypelist[
> isotope_Entity] := {(IsotopeData[isotope, "Name"] \[DirectedEdge]
> IsotopeData[ #[[1]], "Name"]) -> Drop[#, 1]} & /@
> Select[Transpose[{IsotopeData[isotope, "DaughterNuclides"],
> PercentForm /@ IsotopeData[isotope, "BranchingRatios"],
> IsotopeData[isotope, "DecayModeSymbols"],
> IsotopeData[isotope, "DecayEnergies"]}], Not[MissingQ[#[[4]]]] &]
> (* GetEdgeLabel[isotope_Entity]:=Flatten[(decaytypelist[#]&/@children[\
> isotope]),1] *)
> GetEdgeLabel[i1_Entity, i2_Entity] :=
> Block[{dtl = decaytypelist[i1], s2 = IsotopeData[i2, "Name"], s},
> If[Length[dtl] == 0,
> Missing[],
> s = Select[dtl, #[[1, 1, 2]] == s2 &];
> If[Length[s] == 0,
> Missing[],
> Rule[DirectedEdge[i1, i2], s[[1, 1]]]
> ]
> ]
> ]
> makeEdgeLabels[isotope_Entity] := Block[{chil = children[isotope]},
> (* very inefficient, because decay needs only Z1 > Z2 instead auf Outer
> *)
> Flatten[DeleteMissing[Outer[GetEdgeLabel, chil, chil], 2]]
> ]
>
>
> GetEdgeLabel[] ist natürlich auszukommentieren, und
>
>
> Clear[makeDecayGraphSample]
> makeDecayGraphSample[isotope_Entity] :=
> RelationGraph[DaughterNuclidesQ, children[isotope],
> VertexLabels -> makeVertexLabels[isotope],
> EdgeLabels -> makeEdgeLabels[isotope],
> PlotRangePadding -> 0.65, ImageSize -> 300, PlotTheme -> "Scientific"
> ]
>
>
> erscheint das Bild, das natürlich gerade nicht schön ist, weil die Labels
> viel zu lang sind ... go ahead to make it appealing
>
>
> grüsse
>
> Udo.-
>
>
>
>
>
>
>
> Am 23.08.2020 um 19:25 schrieb Peter Klamser:
>
> Lieber Udo,
> zuerst herzlichen Dank für die sehr gute Hilfe.
> Ich hatte mich aber inzwischen weiter durchgekämpft.
> dabei bin ich weiter gekommen, aber bei EdgeLabel lauf ich gegen eine Wand.
> WRI schreibt in der Hilfe:
> [image: image.png]
> Deswegen habe ich die Funktion
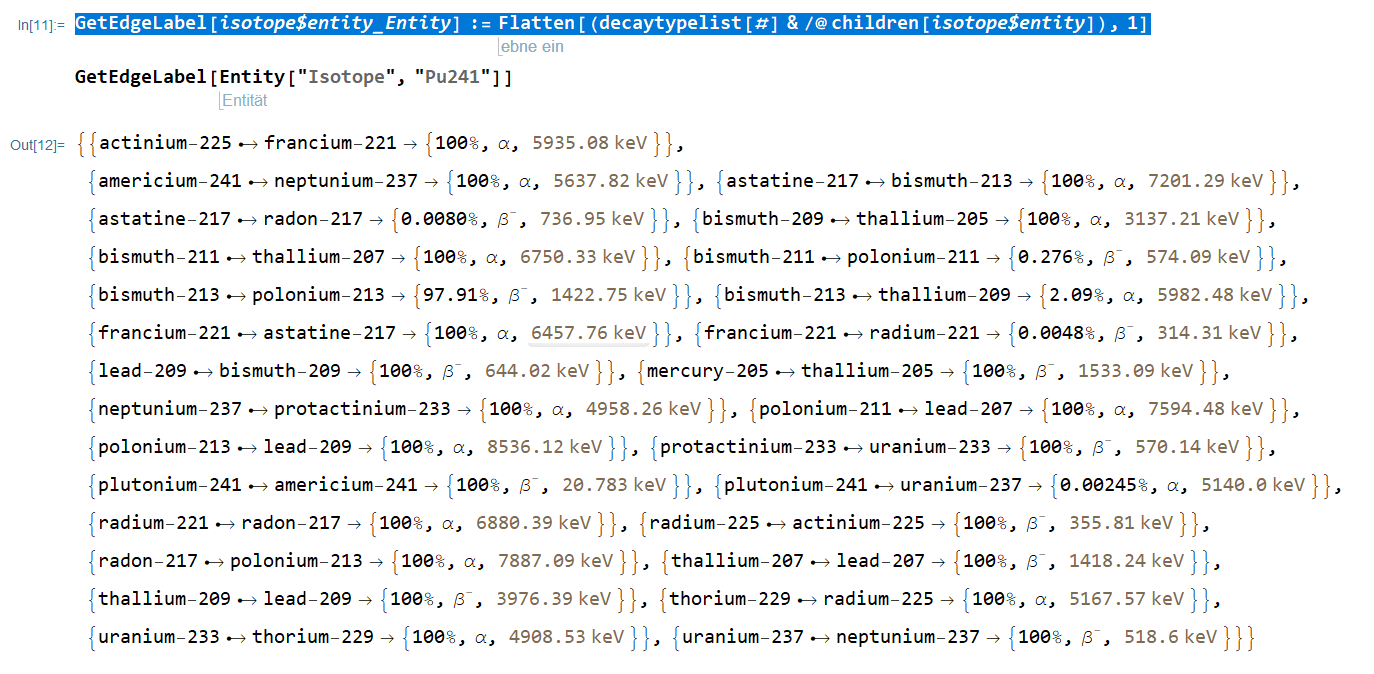
> GetEdgeLabel[isotope$entity_Entity] :=
> Flatten[(decaytypelist[#] & /@ children[isotope$entity]), 1]
> entworfen, die das erwartete Ergebnis liefert:
> [image: image.png]
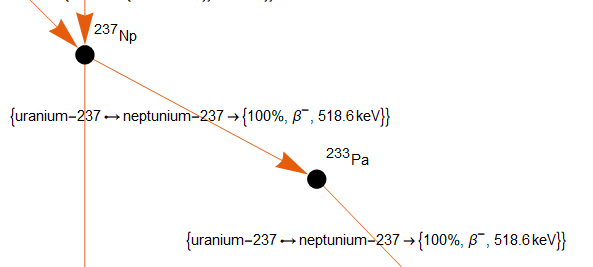
> Wenn ich das ausführe
>
> makeDecayGraphSample[isotope$entity_Entity] :=
> RelationGraph[DaughterNuclidesQ, children[isotope$entity],
> Sequence[VertexLabels -> makeVertexLabels[isotope$entity],
> PlotRangePadding -> 0.65, ImageSize -> 300,
> PlotTheme -> "Scientific"](*,EdgeLabels\[Rule]"Index"*),
> EdgeLabels -> GetEdgeLabel[isotope$entity]];
> (*makeDecayGraphSample[#]&/@Table[Entity["Isotope","Pu"<>ToString[n]],\
> {n,241,241}]//MatrixForm*)
> makeDecayGraphSample[Entity["Isotope", "Pu241"]]
>
> dann wird immer wieder das Ergebnis für nur ein Iotop bei allen Isotopen
> im RelationGraph ausgegeben:
> [image: image.png]
> Danke und eine gute Woche wünscht Peter
>
> Am Sa., 22. Aug. 2020 um 18:21 Uhr schrieb Susanne & Udo Krause <
> su.krause@XXXXXXX.ch>:
>
>> Hallo Peter,
>>
>> zu den Funktionen, die schon seit dem letzten Mal (September 2019) da
>> sind, schreibt man hinzu
>>
>>
>> betaDecayQ[s1_Entity, s2_Entity] :=
>> daughterNuclidesQ[s1, s2] &&
>> IsotopeData[s1, "MassNumber"] == IsotopeData[s2, "MassNumber"] &&
>> IsotopeData[s1, "AtomicNumber"] - IsotopeData[s2, "AtomicNumber"] == -1
>>
>> (* beta decay edge selector *)
>> Clear[betaEdge, betaDecay]
>> betaEdge[s1_Entity, s2_Entity] := If[betaDecayQ[s1, s2],
>> {s1, s2},(* else *)
>> Missing[]
>> ]
>>
>> betaDecay[l_List?VectorQ] :=
>> Block[{res, x1, x2,
>> betaP = IsotopeData[
>> EntityClass["Isotope", "BetaDecay"]] \[Intersection] l},
>> res = If[Length[Cases[betaP, _Entity]] == 0,
>> Print["betaDecay::given entities do not emit electrons"];
>> {}, (* else *)
>> DeleteMissing[Flatten[Outer[betaEdge, betaP, l], 1]]
>> ]; (*
>> If you do not use DirectedEdge you catch unintelligible errors \
>> until you do! *)
>> res //. {x1_Entity, x2_Entity} :>
>> Rule[DirectedEdge[x1, x2], {Thick, Blue}]
>> ] /; Length[Cases[l, _Entity]] > 0
>>
>>
>> und erzeugt das Bildchen in der Beilage mit
>>
>>
>> In[168]:= With[{x = vertsPu241},
>> With[{he = alphaDecay[x], el = betaDecay[x]},
>> RelationGraph[daughterNuclidesQ, x,
>> VertexLabels -> (label1 /@ x),
>> EdgeStyle -> Union[he, el],
>> EdgeLabels ->
>> Union[ReplaceAll[he, {Thickness[Large], RGBColor[1, 0, 0]} ->
>> "\[Alpha]"],
>> ReplaceAll[el, {Thickness[Large], RGBColor[0, 0, 1]} ->
>> "\!\(\*SuperscriptBox[\(\[Beta]\), \(-\)]\)"]],
>> PlotRangePadding -> 0.85, ImageSize -> 500,
>> PlotTheme -> "Scientific"]
>> ]
>> ]
>>
>>
>> Bei den restlichen Zerfällen verfahren Sie analog, nachdem Sie die
>> Zerfallsart nachgeschlagen haben.
>>
>> Bei der Zerfallsart sind die Wolfram Curated Data wieder unglaublich
>> kenntnisreich:
>>
>> In[1]:= IsotopeData["Classes"] // Shallow
>>
>> Out[1]//Shallow= {EntityClass["Isotope", "AlphaEmission"],
>> EntityClass["Isotope", "BetaDecay"],
>> EntityClass["Isotope", "BetaDelayedAlphaEmission"],
>> EntityClass["Isotope", "BetaDelayedDeuteronEmission"],
>> EntityClass["Isotope", "BetaDelayedFission"],
>> EntityClass["Isotope", "BetaDelayedFourNeutronEmission"],
>> EntityClass["Isotope", "BetaDelayedNeutronAlphaEmission"],
>> EntityClass["Isotope", "BetaDelayedNeutronEmission"],
>> EntityClass["Isotope", "BetaDelayedThreeNeutronEmission"],
>> EntityClass["Isotope", "BetaDelayedTritonEmission"], <<37>>}
>>
>>
>> Mit den besten Grüssen
>>
>> udo.
>>
>> P.S. 1: Die EdgeLabels erscheinen wirklich klein, man kann die noch
>> verschieben etc. etc. ...
>>
>> P.S. 2: Wenn Sie es mit dem Output der Wolfram Summer School 2019 machen
>> wollen, müssen Sie halt dort eine Funktion EdgeLabels ->
>> makeEdgeLabel[isotope] einfügen.
>>
>>
>> Am 21.08.2020 um 17:04 schrieb Peter Klamser via demug:
>>
>> Hallo, kann mir bitte jemand weiter helfen?
>> Ich will an einem RelationGraph mit einer Zerfallskette die Linie mit der
>> Zerfallsart beschriften.
>> Es gibt da die Funktion EdgeLabels. Aber da komme ich nicht weiter.
>> Das Notebook stammt von:
>> https://www.wolframcloud.com/objects/nbarch/2019/07/2019-07-5kp1y6b/2019-07-5kp1y6b.nb
>>
>> Dort will ich in dem Ausdruck ein Edgelabel hinzufügen:
>>
>> makeDecayGraphSample[isotope_] :=
>> RelationGraph[DaughterNuclidesQ, children[isotope],
>> Sequence[VertexLabels -> makeVertexLabels[isotope],
>> PlotRangePadding -> 0.65, ImageSize -> 300,
>> PlotTheme -> "Scientific"(*,
>> EdgeLabels\[Rule]{3\[UndirectedEdge]1->}*)]]
>> makeDecayGraphSample[Entity["Isotope", "Pu241"]]
>>
>> Danke sagt Peter
>>
>>
>> _______________________________________________
>> DMUG Deutschsprachiges Mathematica-Forum demug@mathematica.chhttp://www.mathematica.ch/mailman/listinfo/demug
>> Archiv: http://www.mathematica.ch/archiv.html
>>
>>


_______________________________________________
DMUG Deutschsprachiges Mathematica-Forum demug@XXXXXXX.ch
http://www.mathematica.ch/mailman/listinfo/demug
Archiv: http://www.mathematica.ch/archiv.html




